Cell Meter 荧光法细胞周期检测试剂盒 红色荧光 适合于流式细胞检测
| Ex (nm) | 537 | Em (nm) | 618 |
| 分子量 | - | 溶剂 | - |
| 存储条件 | - |
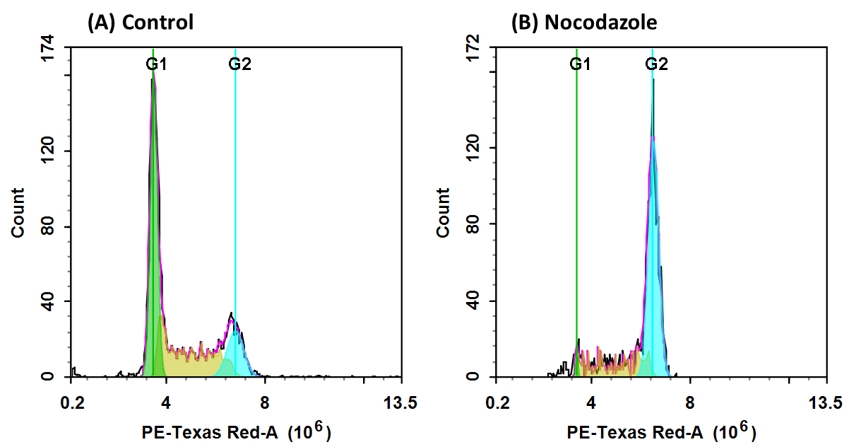
我们的Cell Meter 检测试剂盒是一套用于检测细胞活力和增殖的工具。有多种参数可用于监测细胞活力和增殖。在正常细胞中,DNA密度根据细胞的生长,分裂,静止执行其正常功能而变化。细胞周期的进程受各种细胞周期调节因子之间复杂的相互作用控制。这些调节剂转录因子,该转录因子与DNA结合并开启或关闭导致细胞分裂的蛋白质的产生。这种调节级联中的任何失误都会导致异常细胞增殖,这是许多病理状况(例如肿瘤形成)的基础。活细胞研究的潜在应用是确定细胞DNA含量和细胞周期分布,以检测生长模式的变化,检测凋亡,评估肿瘤细胞的行为和抑制基因的机制。该特定试剂盒旨在使用Nuclear Red CCS1(固定细胞中的细胞周期染色剂)检测细胞周期进程和增殖。染料穿过透化的膜并插入细胞DNA中。试剂盒中提供的RNase降解RNA后,Nuclear Red CCS1的信号强度与DNA含量成正比。可以使用流式细胞仪(FL2通道)检测给定样品中处于G0 / G1,S和G2 / M期的细胞百分比,以及处于凋亡之前的sub-G1期的细胞百分比。百萤生物是AAT Bioquest的中国代理商,为您提供优质的Cell Meter 荧光法细胞周期检测试剂盒。
适用仪器
| 流式细胞仪 | |
| Ex: | 488 nm |
| Em: | 610/20 nm |
| 通道: | PE-Texas Red 通道 |
样品实验方案
简要概述
- 用5×105至1×106cell/ mL的密度制备含测试化合物的细胞
- 用70%乙醇固定细胞
- 将0.5 µL 100X Nuclear Red CCS1和5 µL RNase A加入0.5 mL细胞溶液中
- 在室温下孵育30-60分钟
- 使用带有FL2通道的流式细胞仪分析细胞
实验步骤
1.用测试化合物处理细胞,以诱导凋亡或其他细胞周期功能。
2.对于每个样品,用0.5 mL PBS制备细胞,密度为5×105至1×106细胞/ mL。
2.1对于贴壁细胞:将细胞用胰蛋白酶消化,悬浮在10%FBS培养基中,离心(1000 rpm,5分钟),然后将沉淀重悬于PBS中。
2.2对于悬浮细胞:将细胞离心(1000 rpm,5分钟),并将沉淀物悬浮在PBS(1 mL)中。 注意:应单独评估每种细胞系,以确定诱导凋亡的细胞密度。
3.用70%乙醇固定细胞:
3.1将0.5 mL细胞悬液吸移至1.2 mL无水乙醇中(浓度约为70%)。
3.2在冰上孵育细胞至少2小时(或在-20°C过夜)。在染色之前,细胞可以在-20°C下保存2年。注意:在用单克隆抗体染色细胞表面抗原后,通常将乙醇用于固定,而在用单克隆抗体染色细胞内抗原后,通常将甲醇用于固定。在此过程中,将固定并分析整个细胞。由于仍然存在整个细胞团,因此通常包括使用RNase来任何双链RNA。尽管已经对整个细胞进行了分析,但尝试检测与DNA结合的某些细胞内抗原的尝试可能会失败,因为蛋白质会从透化的细胞中漏出(例如绿色荧光蛋白)。在这些情况下,在酒精固定之前,用PBS中的1%低聚甲醛进行短暂的预固定(在4-6°C下10分钟),将有助于将蛋白质保留在细胞中。
4.用Nuclear Red CCS1染色细胞:
4.1以1000 rpm的速度沉淀细胞5分钟,并用冷PBS至少洗涤一次。
4.2将沉淀物悬浮在0.5 mL测定缓冲液(组分C)中。
4.3加入5 µL 100X Nuclear Red CCS1(组分A)和5 µL 100X RNase A(组分B)。
4.4在室温下孵育细胞30至60分钟。 注意:适当的孵育时间取决于所用的单个细胞类型和细胞浓度。 优化每个实验的孵育时间。
4.5可选:将细胞以1000 rpm离心5分钟,然后将细胞重悬于0.5 mL检测缓冲液(组分B)或自备缓冲液中。
4.6使用FL2通道(Ex / Em = 490/620 nm),通过流式细胞仪检测荧光强度。
参考文献
Cell cycle synchronization of Escherichia coli using the stringent response, with fluorescence labeling assays for DNA content and replication
Authors: Ferullo DJ, Cooper DL, Moore HR, Lovett ST.
Journal: Methods (2009): 8
DNA replication, cell cycle progression and the targeted gene repair reaction
Authors: Engstrom JU, Kmiec EB.
Journal: Cell Cycle (2008): 1402
Morin inhibits the growth of human leukemia HL-60 cells via cell cycle arrest and induction of apoptosis through mitochondria dependent pathway
Authors: Kuo HM, Chang LS, Lin YL, Lu HF, Yang JS, Lee JH, Chung JG.
Journal: Anticancer Res (2007): 395
Direct control of cell cycle gene expression by proto-oncogene product ACTR, and its autoregulation underlies its transforming activity
Authors: Louie MC, Revenko AS, Zou JX, Yao J, Chen HW.
Journal: Mol Cell Biol (2006): 3810
Cell cycle markers for live cell analyses
Authors: Easwaran HP, Leonhardt H, Cardoso MC.
Journal: Cell Cycle (2005): 453
Dynamic relocalization of hOGG1 during the cell cycle is disrupted in cells harbouring the hOGG1-Cys326 polymorphic variant
Authors: Luna L, Rolseth V, Hildrestr and GA, Otterlei M, Dantzer F, Bjoras M, Seeberg E.
Journal: Nucleic Acids Res (2005): 1813
Dynamics of relative chromosome position during the cell cycle
Authors: Essers J, van Cappellen WA, Theil AF, van Drunen E, Jaspers NG, Hoeijmakers JH, Wyman C, Vermeulen W, Kanaar R.
Journal: Mol Biol Cell (2005): 769
Cell cycle regulation of the murine 8-oxoguanine DNA glycosylase (mOGG1): mOGG1 associates with microtubules during interphase and mitosis
Authors: Conlon KA, Zharkov DO, Berrios M.
Journal: DNA Repair (Amst) (2004): 1601
Description of a flow cytometry approach based on SYBR-14 staining for the assessment of DNA content, cell cycle analysis, and sorting of living normal and neoplastic cells
Authors: Nunez R, Garay N, Villafane C, Bruno A, Lindgren V.
Journal: Exp Mol Pathol (2004): 29
Differential roles of STAT1alpha and STAT1beta in fludarabine-induced cell cycle arrest and apoptosis in human B cells
Authors: Baran-Marszak F, Feuillard J, Najjar I, Le Clorennec C, Bechet JM, Dusanter-Fourt I, Bornkamm GW, Raphael M, Fagard R.
Journal: Blood (2004): 2475